Virus ARN bicatenario
| Virus ARN bicatenario | ||
|---|---|---|
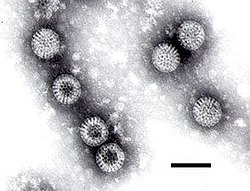 Micrografía electrónica de un rotavirus. La barra mide 100 nm. | ||
| Taxonomía | ||
| Clasificación de Baltimore | ||
| Grupo: | III (Virus ARN bicatenario) | |
Un virus ARN bicatenario (abreviado virus ARNbc o virus dsRNA en inglés) es un virus que tiene como material genético ARN de cadena doble y no se replica usando ADN intermedio. Pertenecen al Grupo III de la Clasificación de Baltimore.[1][2] Como la mayoría de los virus ARN, se replican en el citoplasma y no dependen de las polimerasas de las células huésped como lo hacen los virus ADN, pues incluyen estas enzimas en el virión.
Multiplicación[editar]
Estos virus incluyen una ARN polimerasa dependiente del ARN en el virión, que realiza la transcripción del ARN bicatenario en ARNm.
| Síntesis de proteínas: | ARNbc → ARNm → proteínas |
| Replicación del genoma: | ARNbc → ARNmc- → ARNbc |
| Enzimas: | ARN polimerasas aportadas o codificadas por el virus: ARNbc → ARNm, ARNbc → ARNmc+, ARNmc+ → ARNbc |
La multiplicación del virus comprende las siguientes etapas:

- Transcripción primaria del ARN bicatenario dentro del virión usando la ARN polimerasa dependiente del ARN viral y liberación del ARN monocatenario positivo (que tiene carácter de ARNm) obtenido en el citoplasma. La ARN polimerasa, además es una proteína estructural, ya que forma parte de la cápsida, por ello el virus sólo se replica si a la célula entra la cápsida junto con el genoma vírico.
- Traducción del ARNm, obtención y acumulación de las proteínas virales estructurales y reguladoras.
- Ensamblado parcial del ARN monocatenario positivo y las proteínas virales en viriones inmaduros.
- Transcripción del ARN monocatenario positivo a ARN bicatenario dentro de los viriones por la ARN polimerasa dependiente del ARN viral.
- Transcripción secundaria del ARN bicatenario.
- Ensamblado final y maduración de los viriones.
Los genomas suelen ser segmentados: pueden estar formados por un solo segmento (Totiviridae), dos (Birnaviridae y Partitiviridae), tres (Cystoviridae) o más (Reoviridae con 10-12 segmentos). La replicación suele ser monocistrónica, lo que significa que cada uno de los segmentos codifica una sola proteína, a diferencia de otros virus que exhiben una traducción más compleja. El tamaño del genoma está comprendido entre 4 y 27 kpb. Una característica que distingue a los virus ARN bicatenarios, independentemente de la familia a la que pertenezcan, es su capacidad para llevar a cabo la transcripción de los segmentos de ARN bicatenarios bajo las condiciones apropiadas dentro de la cápsida. En todos estos virus, las enzimas requeridas para la transcripción endógena son, por tanto, parte de la estructura del virión.[3]
También varían en el número de segmentos del genoma (uno a doce) y en la organización del virión (número T, capas de la cápsida y espículas).
Por huéspedes, ejemplos de taxones que infectan hongos son las familias Chrysoviridae, Quadriviridae, Hypoviridae, Megabirnaviridae, Curvulaviridae, en bacterias; Cystoviridae, Picobirnaviridae y el filo Artimaviricota, en animales; Birnaviridae. Otros taxones en cambio infectan huéspedes distintos por cruzado, protistas y hongos; Totiviridae, Polymycoviridae, Fusariviridae, plantas y hongos; Amalgaviridae, plantas, protistas, hongos y bacterias; Partitiviridae, animales, plantas, hongos y protistas; Reoviridae, todo tipo de microorganismos; el filo Taraviricota.
Como es de ver las familias Reoviridae y Partitiviridae son las más extensas y diversas en términos del rango de huéspedes. En la familia Reoviridae se incluyen los Rotavirus, la causa más común de gastroenteritis en niños pequeños en todo el mundo, y el virus de la lengua azul,[4][5] un patógeno de vacas y ovejas de gran importancia económica.
En los últimos años se han logrado importantes avances en la determinación, a niveles atómico y subnanométrico, de la estructura de varias proteínas virales clave y de la cápsida en varios virus ARN bicatenarios. Son destacables las similitudes que exhiben muchos de estos virus en cuanto a su estructura y procesos de replicación. El conocimiento detallado de los aspectos fundamentales de las relaciones entre la estructura y las funciones del virus, ensamblado de la partícula de virus, interacciones virus-célula y de la patogénesis viral permirá el desarrollo de nuevas estrategias y agentes antivirales.[3]
Clasificación taxonómica[editar]
La clasificación taxonómica del ICTV y complementada con otros estudios es la siguiente:[6][7]
- Filo Duplornaviricota
- Filo Artimaviricota
- Filo Taraviricota (en parte)
- Filo Pisuviricota (en parte)
- Clase Duplopiviricetes
- Familia Birnaviridae[8]
- Familia Polymycoviridae[9]
Véase también[editar]
Referencias[editar]
- ↑ "ICTVdb Index of Viruses: Virus Taxonomy, 8th Reports of the International Committee on Taxonomy of Viruses: Listing in Taxonomic Order." (Website). U.S. National Center for Biotechnology Information, National Library for Medicine, National Institutes of Health. Consultado el 09-28-2007.
- ↑ N.J. Dimmock, A.J. Easton y K. Leppard, Introduction to Modern Virology, Ed. Wiley, 2006, ISBN 978-1-4051-3645-7
- ↑ Roy P (2008). «Molecular Dissection of Bluetongue Virus». Animal Viruses: Molecular Biology. Caister Academic Press. ISBN 978-1-904455-22-6.
- ↑ Roy P (2008). «Structure and Function of Bluetongue Virus and its Proteins». Segmented Double-stranded RNA Viruses: Structure and Molecular Biology. Caister Academic Press. ISBN 978-1-904455-21-9.
- ↑ «Virus Taxonomy: 2019 Release» (html). International Committee on Taxonomy of Viruses (ICTV) (en inglés). October 2018. Consultado el 13 octobre 2019.
- ↑ Cryptic and abundant marine viruses at the evolutionary origins of Earth’s RNA virome. Science org.
- ↑ Yuri I. Wolf, Sukrit Silas, Yongjie Wang, Shuang Wu, Michael Bocek, Darius Kazlauskas, Mart Krupovic, Andrew Fire, Valerian V. Dolja & Eugene V. Koonin (2020). Doubling of the known set of RNA viruses by metagenomic analysis of an aquatic virome. Nature.
- ↑ Suvi Sutela, Anna Poimala, Eeva J Vainio (2019). Viruses of fungi and oomycetes in the soil environment. Oxford Academic.
Enlaces externos[editar]
 Wikispecies tiene un artículo sobre Virus ARN bicatenario.
Wikispecies tiene un artículo sobre Virus ARN bicatenario. Wikimedia Commons alberga una categoría multimedia sobre Virus ARN bicatenario.
Wikimedia Commons alberga una categoría multimedia sobre Virus ARN bicatenario.- ViralZone
- Microbiology
- Replication Strategy of dsRNA Viruses
